R语言因子类型的实现
1.因子
R 中用因子代表数据中分类变量 , 如性别、省份、职业。有序因子代表有序量度,如打分结果,疾病严重程度等。
用 factor() 函数把字符型向量转换成因子,如
x <- c(" 男", " 女", " 男", " 男", " 女")
sex <- factor(x)
sex
返回:
![]()
attributes(sex)
返回:

因子有 class 属性,取值为"factor",还有一个 levels(水平值) 属性,此属性可以用 levels() 函数访问,如
levels(sex)
返回:
![]()
因子的 levels 属性可以看成是一个映射,把整数值 1,2, . . . 映射成这些水平值,因子在保存时会保存成整数值 1,2, . . . 等与水平值对应的编号。这样可以节省存储空间,在建模计算的程序中也比较有利于进行数学运算。
事实上,read.csv() 函数的默认操作会把输入文件的字符型列自动转换成因子,这对于性别、职业、地名这样的列是合适的,但是对于姓名、日期、详细地址这样的列则不合适。所以,在 read.csv() 调用中经常加选项 stringsAsFactors=FALSE 选项禁止这样的自动转换,还可以用 colClasses 选项逐个指定每列的类型。
用 as.numeric() 可以把因子转换为纯粹的整数值,如
as.numeric(sex)
返回:
![]()
因为因子实际保存为整数值,所以对因子进行一些字符型操作可能导致错误。用 as.character() 可以把因子转换成原来的字符型,如
as.character(sex)
返回:
![]()
为了对因子执行字符型操作(如取子串),保险的做法是先用 as.character() 函数强制转换为字符型。
factor() 函数的一般形式为
factor(x, levels = sort(unique(x), na.last = TRUE), labels, exclude = NA, ordered = FALSE)
可以用选项 levels 自行指定各水平值 , 不指定时由 x 的不同值来求得。可以用选项 labels 指定各水平的标签 , 不指定时用各水平值的对应字符串。可以用 exclude 选项指定要转换为缺失值 (NA) 的元素值集合。如果指定了 levels, 则当自变量 x 的某个元素等于第 j 个水平值时输出的因子对应元素值取整数 j, 如果该 元素值没有出现在 levels 中则输出的因子对应元素值取NA。 ordered 取真值时表示因子水平是有次序的 (按编码次序)。
在使用 factor() 函数定义因子时,如果知道自变量元素的所有可能取值,应尽可能使用 levels= 参数指定这些不同可能取值,这样,即使某个取值没有出现,此变量代表的含义和频数信息也是完整的。自己指定 levels= 的另一好处是可以按正确的次序显示因子的分类统计值。
因为一个因子的 levels 属性是该因子独有的,所以合并两个因子有可能造成错误,但在最新版本4.1.2 没有报错,应跟版本有关,如
li1 <- factor(c(' 男', ' 女'))
li2 <- factor(c(' 男', ' 男'))
c(li1, li2)
返回:
![]()
如果结果不是因子的话,需要对其进行转换。则那样正确的做法是
factor(c(as.character(li1), as.character(li2)))
就是先转换为字符型,再进行合并就可以了。
2.table() 函数
用 table() 函数统计因子各水平的出现次数(称为频数或频率)。也可以对一般的向量统计每个不同元素的出现次数。如
x <- c(" 男", " 女", " 男", " 男", " 女")
sex <- factor(x)
table(sex)
返回:

对一个变量用 table 函数计数的结果是一个特殊的有元素名的向量,元素名是自变量的不同取值,结果的元素值是对应的频数。单个因子或单个向量的频数结果可以用向量的下标访问方法取出单个频数或若干个频数的子集。
3.tapply() 函数
可以按照因子分组然后每组计算另一变量的概括统计。如
x <- c(" 男", " 女", " 男", " 男", " 女")
sex <- factor(x)
h <- c(165, 170, 168, 172, 159)
tapply(h, sex, mean)
返回:
![]()
4.forcats 包的因子函数
如果使用此函数,需要先进行载入:
library(forcats)
在分类变量类数较多时,往往需要对因子水平另外排序、合并等,forcats 包提 供了一些针对因子的方便函数。
orcats::fac_reorder() 可以根据不同因子水平分成的组中另一数值型变 量的统计量值排序。如:
set.seed(1)
fac <- sample(c("red", "green", "blue"), 30, replace=TRUE)
fac <- factor(fac, levels=c("red", "green", "blue"))
x <- round(100*(10+rt(30,2)))
res1 <- tapply(x, fac, sd); res1
返回:
![]()
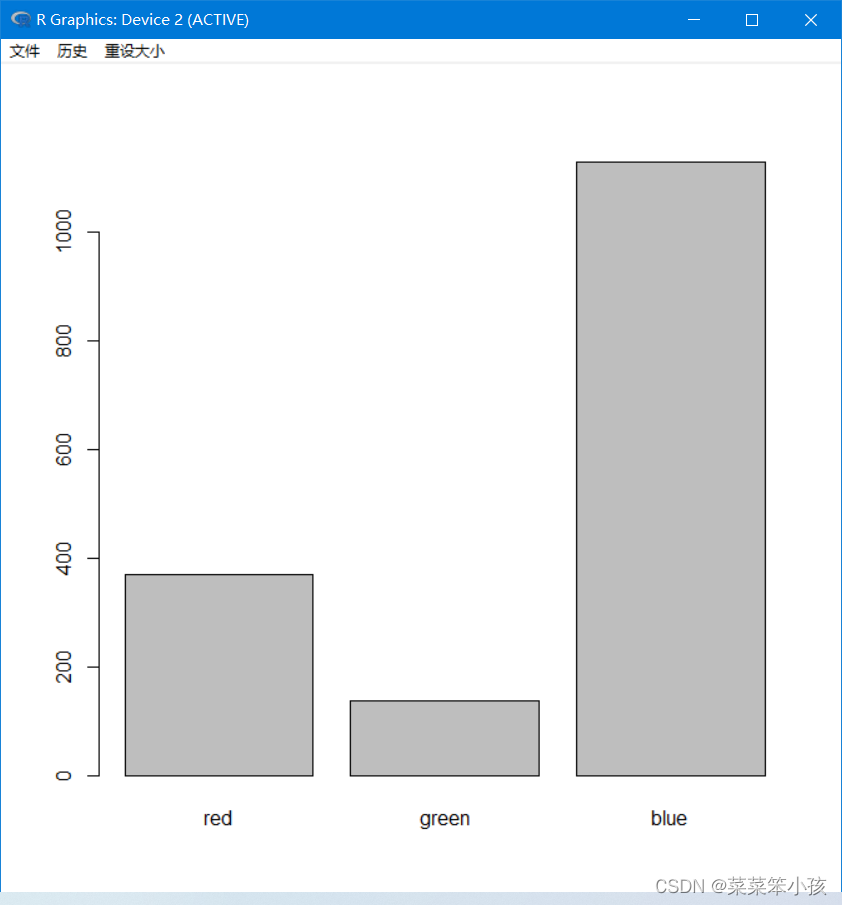
对上面数值画条形图:
barplot(res1)
返回:
如果希望按照统计量次序对因子排序,可以用 forcats::fct_reorder() 函数,并画图条形图,如
fac2 <- fct_reorder(fac, x, sd) res2 <- tapply(x, fac2, sd) barplot(res2)
返回:
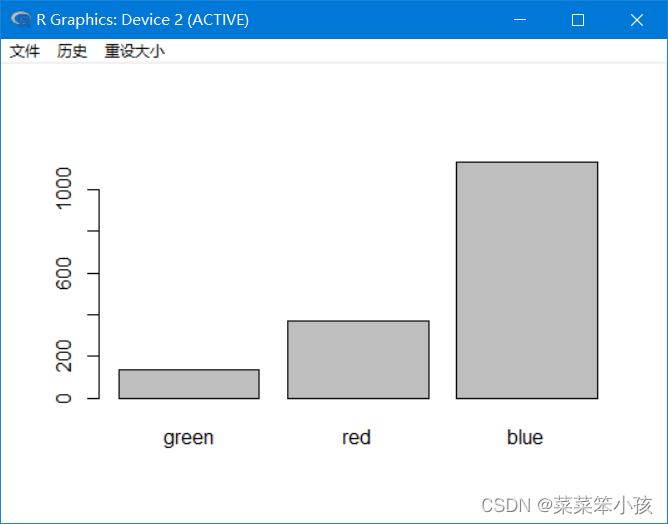
新的因子 fac2 的因子水平次序已经按照变量 x 的标准差从小到大排列。
有时在因子水平数较多时仅想将特定的一个或几个水平次序放到因子水平最前面,可以用 forcats::fct_relevel() 函数,如:
levels(fac)
返回:
![]()
fac3 <- fct_relevel(fac, "blue"); levels(fac3)
返回:
![]()
fct_relevel() 第一个参数是要修改次序的因子,后续可以有多个字符型参数表示要提前的水平。
forcats::fct_reorder2(f, x, y) 也调整因子 f 的水平的次序,但是根据与每组中最大的 x 值相对应的 y 值大小调整次序,这样在作多个因子水平对应的曲线图时可以比较容易地区分多条曲线。
forcats::fct_recode() 可以修改每个水平的名称,如:
fac4 <- fct_recode( fac, " 红"="red", " 绿"="green", " 蓝"="blue") table(fac4)
返回:

fct_recode() 在修改水平名时允许多个旧水平对应到一个新水平,从而合并原来的水平。如果合并很多,可以用 fct_collapse() 函数,记得要先导入forcats 包的因子函数,如
compf <- fct_collapse(
comp,
" 其它"=c("", " 无名", " 无应答"),
" 联想"=c(" 联想", " 联想集团"),
" 百度"=c(" 百度", " 百度集团") )
如果某个因子频数少的水平很多,在统计时有过多水平不易展示主要的类别,可以用 forcats::fct_lump(f) 合并,缺省地从最少的类合并一直到 “ 其它 ” 类超过其它最小的类之前,可以用 n= 参数指定要保留多少个类。
练习
设文件 class.csv 中包含如下内容 :
name,sex,age,height,weight
Alice,F,13,56.5,84
Becka,F,13,65.3,98
Gail,F,14,64.3,90
Karen,F,12,56.3,77
Kathy,F,12,59.8,84.5
Mary,F,15,66.5,112
Sandy,F,11,51.3,50.5
Sharon,F,15,62.5,112.5
Tammy,F,14,62.8,102.5
Alfred,M,14,69,112.5
Duke,M,14,63.5,102.5
Guido,M,15,67,133
James,M,12,57.3,83
Jeffrey,M,13,62.5,84
John,M,12,59,99.5
Philip,M,16,72,150
Robert,M,12,64.8,128
Thomas,M,11,57.5,85
William,M,15,66.5,112
用如下程序把该文件读入为 R 数据框 d.class, 其中的 sex 列已经自动转换为因
子。取出其中的 sex 和 age 列到变量 sex 和 age 中
d.class <- read.csv('class.csv', header=TRUE)
sex <- d.class[,'sex']
age <- d.class[,'age']
(1) 统计并显示列出 sex 的不同值频数;
(2) 分男女两组分别求年龄最大值;
(3) 把 sex 变量转换为一个新的因子, F 显示成 “Female” , M 显示成 “Male” 。
关于R语言因子类型的实现的文章就介绍至此,更多相关R语言因子类型内容请搜索编程宝库以前的文章,希望以后支持编程宝库!
1. W检验(Shapiro–Wilk (夏皮罗–威克尔 ) W统计量检验)目标:检验数据是否符合某正态分布,如:标准正态分布N(0,1)R函数:shapiro.test().结果含义:当p ...